


|

|
|
|
Das EU-Projekt Data GridEurovisionMarcus Hardt |

|
Am Europäischen Kernforschungszentrum Cern in Genf befindet sich zur Zeit der Large Hadron Collider (LHC) im Bau, ein Teilchenbeschleuniger nie da gewesenen Ausmaßes. An vier Stellen des Beschleunigerrings sind Experimente geplant, die ab 2007 die Zerfallsprodukte der Kollision von Protonen oder schweren Ionen aufzeichnen sollen. Die Genauigkeit der Detektoren ist so hoch, dass jeder im Schnitt 2000 Terabyte Daten pro Jahr produzieren wird - insgesamt rechnet man mit zirka 10 Petabyte aufzuzeichnender Zerfallsereignisse pro Jahr. Vergleichbar ist diese Datenflut mit einem 16 Quadratmeter großen Raum voller DVDs, der jedes Jahr neu gefüllt wird - und das 20 Jahre lang.
Die Zerfallsereignisse müssen aber nicht nur gespeichert werden, Wissenschaftler suchen in ihnen auch nach Signaturen von Elementarteilchen wie dem Higgs. Es ist eines der letzten Bausteine in einer Theorie, die ein umfassendes physikalisches Verständnis unserer Welt verspricht. Die Suche nach dem Higgs ist schwer, weil unter den Wissenschaftlern wenig Einigkeit über die Eigenschaften dieses Teilchens besteht. Die berühmte Suche nach der Nadel im Heuhaufen ist ein Kinderspiel dagegen.
Die Suche nach dem Higgs und anderen physikalischen Phänomenen führen Tausende von Physikern weltweit parallel durch und jeder von ihnen möchte den gesamten gespeicherten Datensatz zu seiner uneingeschränkten Verfügung haben. Hieraus ergibt sich ein enormer Rechenzeitbedarf mit der zusätzlichen Komplikation, dass selbst eine zentrale Speicherung der Daten aufgrund ihres Umfangs schwierig ist.
Man könnte annehmen, dass die Hardware eines Tages schnell genug sein wird und genug Speicherkapazität bereitstellt, damit dieses Problem lösbar ist. Allerdings wird eine steigende Anzahl von Nutzern auf wachsende Datenmengen (rund 10 PByte pro Jahr) zugreifen. Daher dürfte der CPU-Bedarf des LHC schneller wachsen, als die von Moores Gesetz vorausgesagte, alle 18 Monate erfolgende Verdopplung der Computerleistung auffangen kann. Das Problem der verteilten Datenanalyse tritt aber nicht nur bei der Teilchenphysik auf. Neben der Hochenergiephysik kommt auch die Biologie bald nicht mehr ohne Grid-Technologien aus. Ein weiteres Anwendungsfeld ist die Analyse von Daten der Erdbeobachtung.
Was also tun? Das von der Europäischen Union finanzierte Projekt European Data Grid (EDG) [1] beschäftigt sich mit Methoden, die Daten und die darauf zugreifenden Rechenjobs zu verteilen. Die Projektmitarbeiter entwickeln eine Infrastruktur, die dem Anwender einen möglichst transparenten Zugang hierauf verschafft. Gleichzeitig eingeführte Optimierungen erlauben es zudem, Jobs zu den Daten zu schicken oder häufig benötigte Datensätze dupliziert vorzuhalten.
Die Komponenten des European Data Grid bauen auf der Version 2 des Globus-Toolkits[2] auf. Weltweit verteilt existieren bereits große Rechenzentren, deren Ressourcen teilweise brachliegen. Viele betreiben Linux-Cluster und stellen Massenspeicher zur Verfügung. Quantitativ reichen diese Zentren aber noch nicht aus, um die Anforderungen des LHC zu erfüllen. Zur Entwicklung einer Grid Middleware - des Software-Layers, der das Rechnen im Grid ermöglicht - reichen aber auch kleinere Aufbauten zunächst völlig aus.
Zurzeit sind im EDG-Entwicklungsbereich etwa 15 Rechenzentren beteiligt. Sie stellen jeweils zwischen zwei und 32 CPUs und bis zu 1 Terabyte Massenspeicher für Tests zur Verfügung. Jedes dieser Rechenzentren stellt ein Computing Element (CE) auf, das Jobs annimmt und an die dahinter liegenden Arbeitsknoten (WN, Worker Nodes) weiterleitet, die die Jobs schließlich ausführen. Hierbei kommen vor allem Komponenten des Globus-Toolkits zum Einsatz (siehe Artikel in diesem Heft). Die Authentifizierung beruht meist auf der Grid Security Infrastructure (GSI, siehe Artikel in diesem Heft), einem Verfahren, das ein X.509-Zertifikat des Nutzers einem lokalen Account zuordnet.
Um auf Massenspeicher zuzugreifen, stehen den Jobs mehrere Möglichkeiten zur Verfügung. Üblicherweise benutzt ein Cluster ein verteiltes Dateisystem, das es im einfachsten Fall von einem Storage Element (SE) per NFS (Network File System) importiert. Liegen Dateien nicht lokal vor, lädt ein Job sie von einem entfernten SE mit dem Programm Gridftp herunter. Diese erweiterte Implementation des File Transfer Protocol (FTP) ist mittlerweile vom Global Grid Forum (GGF)[3] standardisiert.
Es unterstützt Authentifizierung via GSI (Grid Security Infrastructure), Verschlüsselung mit SSL (Secure Socket Layer) sowie so genannte Third Party Transfers, bei denen ein Nutzer am Standort A eine Datei direkt von B nach C kopiert, ohne sich in einem von beiden Standorten einloggen zu müssen.
Eine über Gridftp hinausgehende Möglichkeit, entfernte Dateien in den lokalen Cluster zu schleusen, ist die Replikation. Dafür hat das EDG-Projekt den Replica-Manager entwickelt. Der Kerngedanke ist es, Dateien in einem Replika-Katalog zu registrieren. So ergibt sich eine Übersicht, wo welche Datei verfügbar ist. Das funktioniert so: Jede Datei, die im Replika-Katalog registriert ist, hat einen physikalischen Dateinamen (PFN, Physical File Name). Er ist wie ein URL (Uniform Ressource Locator) aufgebaut: Der Server, auf dem die Datei gespeichert ist, ist Teil des PFN.
Der Nutzer erhält bei der Registrierung im Replika-Katalog einen logischen Dateinamen (LFN, Logical File Name), mit dem er sich eine Referenz auf die physikalische Datei beschafft. So weist der Nutzer oder einer seiner Jobs den Replika-Manager dazu an, die logische Datei am gewünschten Ort zu replizieren. Dort entsteht eine zweite physikalische Instanz derselben Datei. Das System weiß daher, welche physikalischen Kopien zum gegebenen logischen Dateinamen existieren.
So kann es in Abhängigkeit von Faktoren wie Netzwerkauslastung, Verfügbarkeit und Bandbreite selbst entscheiden, von wo aus es die physikalischen Dateien am besten holt. Die Dateien kopiert der Benutzer dann wieder mit Gridftp. Diese Methode funktioniert aber nur so einfach, wenn es sich um Daten handelt, auf die der Anwender lesend zugreift. Für den Schreibzugriff auf Replika-Daten gibt es bis jetzt nur Konzepte.
Die Zugriffsrechte auf Dateien sind auf die Mitgliedschaft in virtuellen Organisationen abgebildet (Abbildung 1). Das Konzept erleichtert die Verwaltung, da nicht jedes Rechenzentrum jedem Nutzer Accounts einrichten muss. Stattdessen entscheidet eine Zentrale, welche virtuellen Organisationen es zulässt. Technisch entspricht dies der Entscheidung, aus dem Teil eines LDAP-Baums, der der akzeptierten VO zugeordnet ist, die Header von Zertifikaten auszulesen und diesen mit Hilfe der Globus-GSI eine Unix-Group-ID zuzuordnen.
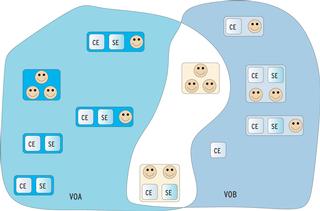
Das Team des EDG hat das Verfahren dahin weiterentwickelt, dass jeder Benutzer einen freien Pool-Account zur Verfügung gestellt bekommt. Damit verhindert es, dass lokale Benutzerkonten inflationär auftreten.
Um an einem Grid teilzunehmen, muss ein Benutzer zunächst Mitglied in einer virtuellen Organisation sein. Dazu importiert er sein Zertifikat in einen Browser und akzeptiert auf der Webseite der virtuellen Organisation die Nutzungsbedingungen. Außerdem ist noch ein Aufnahmeantrag digital zu unterschreiben. Nach der Aufnahme wird sein Zertifikat via LDAP veröffentlicht. Die Mitglieder derselben virtuellen Organisation sind auf den Worker Nodes auch Mitglieder in denselben Gruppen des Linux-Betriebssystems.
Der Information Index (II) ist ein wesentlicher Bestandteil des EDG (Abbildung 2). Er unterstützt die Planung, indem er Auskunft darüber gibt, wo es wie viele Ressourcen gibt und wie viele tatsächlich verfügbar sind. Hier kommt der vom Globus-Projekt entwickelte Meta Directory Service (MDS) zum Einsatz. Grid Resource Information Server (GRIS) sammeln Informationen und leiten sie über eine Hierarchie von Grid Information Index Servern (GIIS) weiter. Diese Informationen stellt schließlich ein LDAP-Baum zur Verfügung.

Die aktuelle Entwicklerversion des EDG setzt bereits die so genannte Relational Grid Monitoring Architecture (RGMA) ein. Diese Eigenentwicklung von Data Grid besteht aus Informations-Qellen, -Konsumenten und -Speichern. Die Informationsspeicher sind herkömmliche relationale MySQL-Datenbanken, verteilt auf die Rechenzentren.
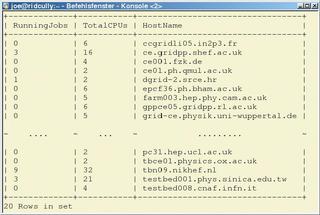
Die Informationsquellen beschaffen die Informationen, beispielsweise aus »/proc/*«. Die Speicher bewahren diese Informationen auf. Das System sorgt selbst dafür, dass sich die Informationen bis zum Konsumenten fortpflanzen. Es gibt neben einer API noch eine Kommandozeilen-Schnittstelle, die sich stark an SQL anlehnt. Beispielsweise liefert ein Kommando wie
latest select RunningJobs,TotalCPUs, HostName from GlueCE
eine Liste aller Computing-Elemente (CE) und beantwortet, wie viele Jobs dort zurzeit laufen und wie viele CPUs verfügbar sind. Abbildung 3 zeigt das Ergebnis. So lässt sich auch feststellen, welche Art von Massenspeicher in welcher Größe verfügbar ist. Auch die Qualität der Verbindungen zwischen den einzelnen Rechenzentren veröffentlicht das Informationssystem permanent.
Vor lauter Ressourcen konnte der Benutzer bisher leicht den Überblick verlieren. Um eine einfache Navigation im verteilten Dateisystem zu erlauben, hat die EDG-Arbeitsgruppe Workload Management System den so genannten Ressource Broker (RB) als Herzstück der EDG-Architektur entwickelt. Er informiert sich beim Informationssystem über den aktuellen Status des Grid und findet zudem heraus, wo sich die physikalischen Kopien eines logischen Dateinamens befinden. Die Informationen verwendet er, um den besten Ort für die Ausführung eines Programms zu ermitteln. An dieser Stelle arbeitet ein so genannter Matchmaker, dessen Funktionalität und Arbeitsweise sich an den Condor Matchmaker anlehnt[4]. Der Benutzer schickt also seine Jobs an den Ressource Broker.
Vorher erstellt er mit der Job Description Language (JDL) eine Datei, die den Job beschreibt (Listing 1). Der Benutzer spezifiziert das Programm, das er ausführen möchte, zusammen mit den passenden Argumenten. Stdout und Stderr sollen in die angegebenen Dateien geschrieben werden. Die Input-Sandbox enthält lokale Dateien, die der Job für die Ausführung benötigt, beispielsweise Konfigurationsdateien. Die Output-Sandbox wiederum enthält die Liste aller Dateien, die nach Programmende wieder zurück zum Benutzer müssen.
| Listing 1: Eine JDL-Datei |
|
01 Executable = "./myexecutable";
02 Arguments = "--config small-file.cfg --data BIG-file.dat";
03 StdOutput = "std.out";
04 StdError = "std.err";
05 InputSandbox = {"small-file.cfg"};
06 OutputSandbox = {"std.out","std.err","run.log"};
07 InputData = {"LF:BIG-file.dat"};
08 ReplicaCatalog = "ldap://rls.example.org:12345/lc=WPsixCollection,rc=WPsixRC,dc=testbed,dc=fzk,dc=de";
10 DataAccessProtocol = {"gridftp"};
|
Bei Input- und Output-Sandbox handelt es sich üblicherweise um kleine Dateien, da der Benutzer sie mit jedem Job mitschicken muss. Große Datensätze hingegen registriert der Replika-Katalog vorher und repliziert sie bei Bedarf in verschiedenen Rechenzentren. Per Input-Data teilt der Benutzer dem Ressource Broker mit, dass der Job in einem der Rechenzentren laufen muss, an dem eine physikalische Instanz von »BIG-file.dat« verfügbar ist.
Mit der Software aus dem European Data Grid lassen sich große Rechenaufgaben geschickt verteilen. Zudem entlastet sie die Benutzer, indem sie herausfindet, welche Ressourcen an welchem Ort am sinnvollsten zu verwenden sind. Darauf aufbauend ist es möglich, Anwendungen zu entwickeln, die sehr viele Programme ins Grid schicken und die Anwender grafisch über den Status dieser Jobs informieren.
Ein Beispiel aus der Biomedizin, das im Prinzip wie die Physik-Analysen bei der Suche nach dem Higgs-Teilchen funktioniert, soll die Verteilung der Rechenaufgaben veranschaulichen. Die Software stammt aus der Arbeitsgruppe für biomedizinische Anwendungen, einem Teil von EDG. Dabei vergleichen Ärzte das hoch aufgelöste Röntgenbild einer Lunge mit den entsprechenden Bildern anderer Patienten. Die Diagnosen ähnlicher Bilder möchten sich die Ärzte dann ansehen. Das Szenario führen Entwickler momentan in Rechenzentren mit simulierten Röntgenbildern durch.
Die Problematik lässt sich mit EDG-Komponenten lösen. Alle Röntgenbilder jedes Patienten registriert der Replika- Katalog in verschiedenen Krankenhäusern. So entsteht eine verteilte Wissensbasis. Die Biomed-Anwendung holt sich beim Start eine Liste logischer Dateinamen aller Röntgenbilder, die der Arzt analysieren will. Für jeden logischen Dateinamen erzeugt sie eine JDL-Datei. Sie enthält zusätzlich noch den logischen Dateinamen des aktuell benutzten Röntgenbilds als Input-Sandbox.
Das Executable ist ein Programm, das zwei Bilder vergleicht, und Arguments sind einfach logische Dateinamen. Die Output-Sandbox enthält als Ergebnis einen Wert, der der Qualität der Übereinstimmung entspricht. Das JDL-File übergibt die Software zusammen mit der Input-Sandbox und dem Executable an den Resource Broker. Erst dieser schickt die Jobs jeweils dorthin, wo die benötigten Vergleichsbilder vorhanden sind, und vergleicht die Bilder schließlich vor Ort.
Die verteilte Lösung skaliert besser als ein zentraler Aufbau, da jedes Krankenhaus selbst alle Ressourcen unterhält, um Daten zu speichern und zu verteilen. Selbstverständlich berücksichtigt das Szenario noch nicht die steigende Anzahl von Nutzern. Allerdings tritt das gleiche Problem auch bei einer zentralen Lösung auf.
Das Team des European Data Grid konnte in der knappen Zeit von drei Jahren und drei Monaten eine beeindruckende Anzahl an Softwarekomponenten zur effizienten Verteilung extrem rechen- und datenintensiver Analyseprogramme entwickeln. Das EDG ist damit eine weitreichende Ergänzung des Globus Framework, die der Wissenschaft und Wirtschaft in und außerhalb Europas zugute kommen wird.
Im März 2004 endete das EDG-Projekt nach erfolgreich abgeschlossener Review durch die EU. Nun übernimmt das EGEE-Projekt[5] die weitere Arbeit. Neben einer Weiterentwicklung der Softwarekomponenten in Richtung Grid- und Webservices (WSRF) sowie des Aufbaus von Grid-Infrastrukturen in Europa will EGEE auch die entwickelten Techniken im Rahmen von Schulungen einem breiteren Publikum zugänglich machen und zudem in neue Anwendungsgebiete vordringen. (jre)
| Infos |
|
[1] Europäisches Data-Grid-Projekt: [http://eu-datagrid.org] [2] Globus Alliance: [http://globus.org] [3] Global Grid Forum: [http://gridforum.org] [4] Condor Matchmaker: [http://www.cs.wisc.edu/condor/] [5] EGEE-Projekt: [http://public.eu-egee.org] |
| Der Autor |
|
Marcus Hardt studierte Physik in Aachen. Seit zwei Jahren arbeitet er im Forschungszentrum Karlsruhe am Institut für Wissenschaftliches Rechnen. Dort betreut er zwei Data-Grid-Installationen im Rahmen des EDG und des EU-Projekts Cross Grid. |